亚马逊AWS官方博客
云上大规模蛋白结构预测最佳实践
 |
背景
蛋白质的三维结构与其生物学功能密切相关,决定了它如何与药物化合物相互作用。了解蛋白质的三维结构可以帮助筛选更适合靶向蛋白质结构的药物化合物,从而显著改进药物开发过程。近年来如 AlphaFold 和 RoseTTAFold 等基于人工智能的蛋白质折叠算法在三维结构预测的准确性方面取得了突破性的提升,可以用来快速构建准确的蛋白质结构模型,已经被广泛应用于药物研发以及生命科学行业的其他领域。然而,运行 AlphaFold 等算法需要大量的 GPU 计算资源以及并发访问 TB 级的存储数据,在数千个候选蛋白质结构预测的规模上如何实现可扩展性的高性能对行业用户来说有很大的挑战。
为了帮助客户更好在云上使用使用 AlphaFold 和 RoseTTAFold 等工具进行蛋白结构预测任务,我们开发了蛋白结构预测工作台(Protein Folding Workbench)解决方案。该方案提供了这是一款集企业级安全资源管理、作业智能调度、结果可视化分析于一体的端到端蛋白质结构预测任务管理平台。本文将以实际的用户场景为例,介绍如何通过 Protein Folding Workbench 在云端实现数百个并发规模的蛋白结构预测任务,以及从中总结出的搭建云上蛋白结构预测作业平台的最佳实践。
Protein Folding Workbench 解决方案简介
Protein Folding Workbench(以下简称 PFW)的主要设计目标是为药物研发科学家等行业用户提供简单易用的云上一体化蛋白结构预测平台。其主要功能包括:
- 作业管理:用户通过界面启动作业,对一个蛋白结构预测作业,可以指定输入的文件,所用的实例类型,分析所用的算法,以及运行参数等等,也可以根据多个输入文件启动批量作业。作业运行期间可以查看作业状态,并在作业结束后在 WebUI 直接对结果三维结构进行可视化分析。
- 资源管理:支持用户通过图形化 UI 的方式上传和管理作业用到的输入文件,提交作业时可以直接引用。
- 环境管理:管理项目以及可以访问项目的用户。用户可分为系统用户以及普通用户,系统用户可以访问项目内的所有资源,普通用户只可以管理自己提交的作业,而不能看到其他用户的作业。不同项目之间的资源是完全隔离的,以实现企业内部的数据访问控制。
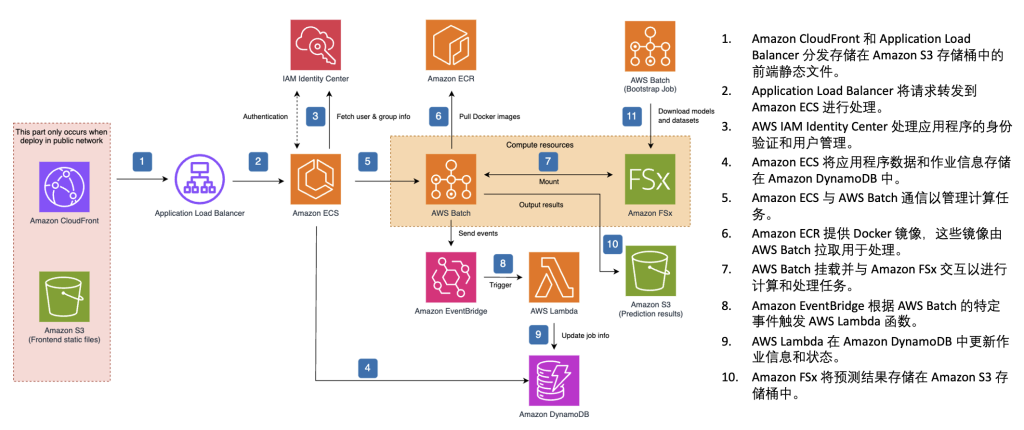 |
图 1. Protein Folding Workbench 参考架构图
如图 1 所示,PFW 方案通过 Amazon CloudFront 与 Application Load Balancer 实现前端静态页面的高效分发与请求转发。用户访问系统后,IAM Identity Center 负责用户身份认证与权限管理。后端服务运行在 Amazon ECS 上,并将应用配置和元数据存储在 Amazon DynamoDB 中。ECS 将推理任务提交至 AWS Batch,后者从 Amazon ECR 拉取容器镜像,加载模型与数据集,并通过 FSx for Lustre 实现高速的读写和临时计算挂载,加速推理计算任务的执行。任务完成后,结果存储在 Amazon S3 中,并通过 Amazon EventBridge 触发 AWS Lambda 函数更新 DynamoDB 中的作业状态信息,从而实现任务状态的自动追踪与异步通知。整体架构具备以下特性:
- 计算与存储分离,使用 FSx for Lustre 提升数据访问性能
- 事件驱动的无服务器编排,基于 EventBridge 与 Lambda 实现任务自动化控制
- 弹性容器化执行,通过 Batch 支持大规模推理作业调度
- 端到端托管服务整合,大幅减少运维开销
整个方案的构建充分考虑到了企业环境的安全要求,所有组件可以部署到用户的 VPC 内,避免将相关数据传输到第三方的服务器。用户通过 IAM Identity Center 管理用户,也可以通过 OIDC 协议对接企业自有的 Identity Provider 服务。通过将用户归属到不同项目,可以实现企业内部不同用户之间的访问权限隔离。另外,所有组件可以部署到 VPC 的私有网络内,避免潜在的来自公网的网络攻击。
客户场景
以下我们将通过一个具体的案例探讨通过 PFW 在云上进行大规模蛋白结构预测工作的最佳实践。生物科技公司A,在药物研发的过程中经常需要在短时间内处理数百个蛋白结构预测作业。客户评估了其他类似 AlphaFold Server 的第三方 SaaS 方案,出于对数据的安全和保密考虑,客户最终选择了将 PFW 部署在自己账号内 VPC 内的方案,这样可以避免将关键数据传输到第三方的服务器上。
初步测试
客户 PoC 的测试目标是在两天内完成 400 个基于 AlphaFold 2 的蛋白结构预测作业。在第一轮测试中,客户使用 PFW 提交 15 个并发运行的 AlphaFold 2 的作业,这些作业的单机运行时间在 1 小时左右。每个作业使用 g4dn.2xlarge 实例,并行文件系统 FSx for Lustre 的存储大小为 2.4TB,默认为单 AZ Scratch 2 模式。测试中最大的并发度为 15 个实例。测试结果发现,15 个作业总体耗时 10 个小时,与单机的运行时间相差很大。而且不同作业之前的完成时间参差不齐,作业的并发性能明显受限。
为了协助排查性能问题,我们分析了 AlphaFold 2 算法,其主要运行阶段如表 1 所示。其中 MSA 与模板搜索运行在 CPU 上,需要检索数百万个输入文件,这些输入文件的大小一般在几个 KB。当多个作业同时运行时,这些并发的小 I/O 会对文件系统产生很大的压力。同时,我们了解到客户优化过的 AlphaFold 算法通过增加 MSA 阶段的并发度来减少运行时间,这会加剧文件系统的 I/O 压力。因为计算实例是相互独立的,我们推测作业的性能问题主要是由于 I/O 瓶颈引起的。
| 阶段名称 | 描述 | 计算资源 |
| MSA 与模板搜索 | 查找进化和结构上下文信息 | CPU |
| 特征嵌入 | 将输入数据进行数值编码 | CPU |
| Evoformer 模块 | 深度学习模型,用于特征学习 | GPU |
| 结构模块 | 预测三维坐标 | GPU |
| 置信度评估 | 预测每个残基和残基对的置信度 | GPU |
表 1. AlphaFold 2 算法的各个阶段
图 2 是测试时段 FSx for Lustre 的运行监控数据。可以看到,客户端总吞吐量在作业开始不久就达到 1.21GB/s,之后逐步降低,并维持在一个较低的水平。值得注意的是 2.4TB 的 FSx for Lustre 提供的标称总吞吐为 480MB/s,1.21GB/s 是 burst 吞吐所达到的最大值。同时,我们可以看到总客户端的 IOPS 基本维持在一个很高的水平,这是因为大量的文件访问都是小 I/O 操作。这些监控数据证实了作业并发导致的 I/O 瓶颈问题。
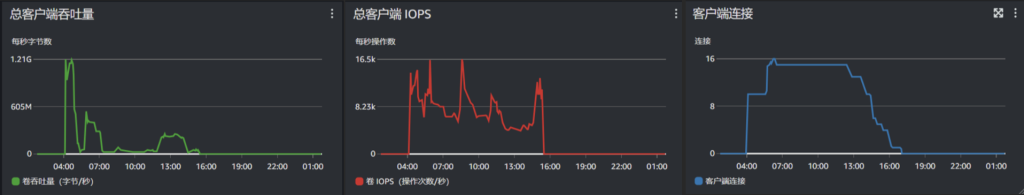 |
图 2. FSx for Lustre 性能监控
另外,针对不同作业之间运行时间差距较大的问题,因为作业需要访问数百万个小文件,而在 PFW 的默认配置中使用的是单可用区的配置,跨可用区访问会产生毫秒级别的延时,所以我们推测跨可用区 I/O 访问的延时是导致性能差别的主要原因。为了验证我们的推测,我们做了个简单的实验,在三个可用区分别用g4dn.2xlarge实例运行了一个序列长度为 500 左右的 AlphaFold 2 作业,其中 FSx for Lustre 部署在可用区 1a。作业运行的结果见表 2,可以看见与文件系统在同一可用区的作业运行时间最短,在可用区 1c 的作业比在可用区 1a 的作业运行时间增加了 74%,验证了我们的推断。
| 机型 | 资源配置 | FSx 容量 | FSx 压缩 | 可用区 | 完成时间 |
| g4dn.2x | 8C 32G | 4800GB | LZ4 | cn-northwest-1a | 02:30 |
| g4dn.2x | 8C 32G | 4800GB | LZ4 | cn-northwest-1b | 03:42 |
| g4dn.2x | 8C 32G | 4800GB | LZ4 | cn-northwest-1c | 04:22 |
表 2. 在不同可用区运行单个 AlphaFold 作业的性能对比
性能优化方案
根据初步测试的结果,我们设计了几个优化的方向:
- 由于作业的瓶颈主要在 I/O,建议增加 FSx for Lustre 的磁盘空间以提升文件系统的 IOPS 和吞吐,通过并发测试找到性能价格比最优的容量配置;
- 同时由于 AlphaFold 算法访问的主要是小文件,计划尝试关掉 FSx for Lustre 的压缩功能观察是否有性能提升;
- 为了避免跨可用区访问文件系统导致的额外的性能损耗,可以把 Batch 的计算实例限制到与 FSx for Lustre 同一个可用区;
根据这些可能的优化方向,我们模拟客户场景设计了一系列的实验比较不同的配置下作业的并发性能。计算实例使用 g4dn.2xlarge 或者 g4dn.4xlarge,作业并发度比较了 10,20,和 50 几个值,FSx for Lustre 文件系统的容量从 4.8TB 逐渐增加到 19.2TB 以观察容量对于作业并发性能的影响。所有的实验均使用原生的 AlphaFold 2 算法,输入文件为 500 字节左右的氨基酸序列。
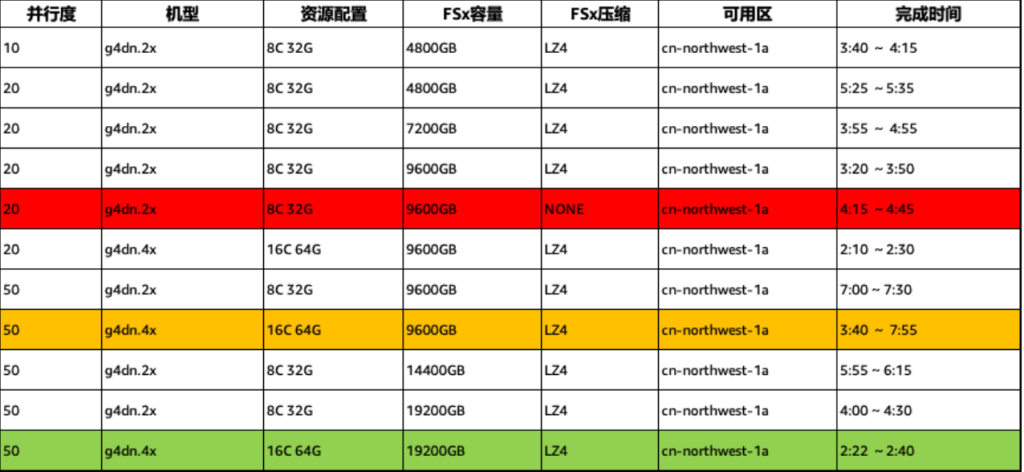 |
表 3. 各种配置下的并发测试性能
各项测试的详细配置以及结果见表 3。从运行时间上看,当作业并发度为 20,计算实例固定为 g4dn.2xlarge 时,随着文件系统容量增大,作业的整体完成时间有明显的降低。在文件系统容量为 9.6TB,把计算实例从 g4dn.2xlarge 替换为 g4dn.4xlarge,作业的运行时间可以进一步降低,这是因为 g4dn.4xlarge 有更多的 vCPU,可以帮助减少 MSA 计算阶段的运行时间。同时,在这个配置下,2.5 小时内可以完成 20 个作业的 batch,1 天内大概可以完成 200 个作业,可以达到客户的 PoC 吞吐要求。如果文件系统的容量提升到 19.2TB,并行运行 50 个 AlphaFold 作业,可以在 2 小时 40 分内完成。这个配置下一天可以完成 400 至 500 个作业。但这个配置下文件系统的容量比较大,因此也会带来更多的方案保有成本。值得一提的是,我们发现关掉 FSx for Lustre 的压缩功能在同样配置下反而导致了性能的下降,因此推荐在生产负载种还是使用默认的文件系统压缩功能。
压力测试
根据以上组合测试的结果,我们推荐在客户的场景下将 FSx for Lustre 的容量设置为 9.6TB 进行压力测试。总作业数为 200,每个 batch 并发运行 20 个作业,实例类型使用 g4dn.2xlarge 和 g4dn.4xlarge 进行比较,以便客户根据成本和效率的需求进行灵活的配置。结果如表 4:
| 实例类型 | 文件系统容量 | 每批任务数 | 总耗时(最后一批任务完成时间) | |
| 配置一 | g4dn.2xlarge | 9.6TB | 20 | 45 小时 ~ 48 小时 |
| 配置二 | g4dn.4xlarge | 9.6TB | 20 | 26 小时 ~ 27 小时 |
表 4. 压力测试耗时结果
可见在压测环境下,系统的吞吐率已经可以完成客户在 2 天内处理 400 个左右作业的 PoC 性能测试要求。
结论
通过本文的实践案例可以看出,利用云上弹性计算和高性能文件系统,可以有效支撑大规模蛋白结构预测任务的并行化和规模化需求。客户通过部署 Protein Folding Workbench(PFW)平台,成功利用云端可扩展架构的提升了预测作业的处理能力和运行效率。在此过程中,优化云上 I/O 性能成为关键因素之一,包括合理配置 FSx for Lustre 文件系统容量、控制计算实例与存储的可用区亲和性等。本文总结出的优化经验和架构最佳实践为未来其他生命科学行业客户在云上部署 AlphaFold 等大规模蛋白结构预测应用提供了重要参考。